长链非编码RNA(lncRNA)是一类不具备蛋白编码能力的RNA,其长度普遍大于200个核苷酸。近七成的人类基因组区域能够进行转录,然而蛋白编码基因只占全部基因组区域的2%左右,其余区域均为非编码区域。早期研究认为这些转录本属于“垃圾”序列,然而近年来越来越多的研究充分证明了lncRNA举足轻重的生物学功能。
与编码蛋白的mRNA敲除策略不同,lncRNA不能通过移码突变进行基因敲除。目前主流的lncRNA敲除方法主要有三种:全基因座敲除、敲入poly(A)终止信号以及敲除启动子和第一外显子(PE1)的方法。然而,这些方法依然存在诸多问题:一,lncRNA与邻近基因位置关系复杂,大范围的DNA片段移除可能导致邻近基因的重要调控序列甚至编码序列破坏,从而扰乱结果观察和分析;二,对于一些多外显子lncRNA可能在第一 外显子下游存在潜在的“备用”启动子序列,从而导致lncRNA敲除失败;三,部分lncRNA存在有多个转录起始位点的现象;四,一些lncRNA中的内含子存在其他ncRNA,5)细胞同源重组修复效率较低。正是由于以上问题,目前lncRNA研究更多是聚焦基因间lncRNA,而与邻近基因有重叠的antisense lncRNA(NAT)等的功能研究相对较少。
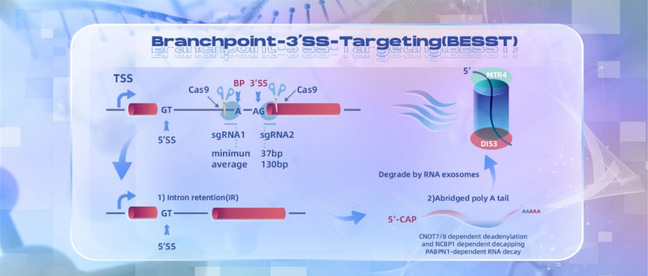
针对此问题,清华大学深圳国际研究生院张雅鸥教授、重庆市人民医院吕青研究员、陆军军医大学曾春雨教授等研究出一种对基因组扰动较小的新式lncRNA敲除策略(BESST),通过一种基于CRISPR-Cas9、将靶lncRNA的最后一个内含子的“分支点”(branchpoint)至3’剪切点(3’splicing site, 3’SS)的DNA片段移除的方法,高效地诱导靶lncRNA核内降解,从而敲除靶lncRNA的功能。
该项研究选取了多条代表性的lncRNA,涵盖了大部分复杂的lncRNA与邻近基因之间的相互位置关系,实验结果显示BESST方法可以有效地敲降这些场景里的lncRNA水平,但并不影响其邻近基因的表达水平,这使得精确解析lncRNA的功能成为可能。此外,本研究还比较了BESST敲除方法与常规的PE1方法,实验结果表明BESST在获得与PE1相似的lncRNA敲降水平的同时对其临近基因以及转录组整体扰动更小。通常应用PE1敲除lncRNA时需要预测并验证启动子位置,普遍需要从染色体上移除数百甚至几千个碱基对,而BESST方法只需要移除~40个碱基对就足以引起整条RNA转录本的彻底降解。
此外,研究团队发现BESST敲除后的lncRNA会滞留在细胞核内。为了进一步研究BESST引起RNA转录本降解的机制,研究人员将细胞核内的核酸降解途径中的主要成分逐一进行敲除,最后发现BESST敲除导致的lncRNA降解是由PABPN1介导的CNOT7/8依赖的脱多聚腺苷化(deadenylation)和NCBP1依赖的脱帽作用导致的。

图1. BESST方法敲除lncRNA的工作示意图
综上,该研究基于CRISPR-Cas9技术,用两条sgRNA切割lncRNA最后一个内含子的分支点上游和3’剪切点的下游,引起靶lncRNA的最后一个内含子的滞留(intron retention),从而导致RNA转录本的核滞留(nuclear retention),最终通过细胞核内的RNA降解途径“消化”靶lncRNA(图1)。该方法的优势体现在:一,敲除区域较短并且“特征”明显,仅需要敲除数十个碱基对就可实现干扰lncRNA的目的;二,对整个基因组的扰动较常规方法显著减少,甚至可以减少对内含子基因的干扰;三,该方法也可应用于蛋白编码基因;四,不干扰lncRNA的正常转录起始。BESST方法的建立为诸多lncRNA的功能研究和体内实验带来新的选择,并可能通过高通量的方式对lncRNA的功能进行快速筛选。
近日,上述成果以“BESST:一种对基因组扰动较小的新式lncRNA敲除策略”(BESST: A novel lncRNA knockout strategy with less genome perturbance)为题发表在《核酸研究》(Nucleic Acids Research)。本文的第一作者是清华大学深圳国际研究生院博士生张世宽,通讯作者是清华大学深圳国际研究生院张雅鸥教授。重庆市人民医院吕青研究员和陆军军医大学曾春雨教授为共同通讯作者,陆军军医大学大坪医院陈悦和佐治亚医学院奥古斯塔大学董坤哲博士为本文的共同第一作者。该研究得到了国家自然科学基金和深圳国际合作项目的资助。
论文链接:https://doi.org/10.1093/nar/gkad197
文/图:吕青
视频:戴雨静
编辑:林洲璐
封面设计:唐慧雯
审核:陈超群


